|
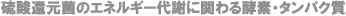



�@ �@�@
�@�@�@���_�Ҍ��ہiSulfate-reducing bacteria�j�́C���_�����ŏI�d�q��e�̂Ƃ��Đ����������c�ޕΐ����C���ۂł���D�܂�C��X���_�f���z���u�_�f�ċz�v�����Ă���̂ɑ��āC���̍ۂ͗��_������荞�݁u���_���ċz�v�����鐶���Ƃ��Ă��D���Â̐́C�_�f�����Ȃ��܂��n�����Ҍ���Ԃł��������ɔɉh���Ă��������݂ł͎_�f�������悤�ɂ��Ēn���ōׁX�Ɛ������тĂ���ƌ����Ă����D����䂦�n���̐����̐i���ɂ��Ă̏���̓��ɋL�^���Ă���Ƃ��l�����邱�Ƃ��炻�̑�Ӌ@�\��^���p�N�����q�̍\�������w�I�����ɋ�����������Ă����D�������C�{���Ɏ_�f���n������ȑO�ɔɉh���Ă��������^�⎋���錤���҂�����D
�@�@�@���������ł�Desulfovibrio vulgaris Miyazaki F�Ƃ������_�Ҍ��ۂ̃G�l���M�[��ӂɏœ_���i���Ă��̍\�������w�I�����𑱂��Ă��Ă���i�ꕔ�CD. v. Hildenborough�Ƃ������������Ă���DHildenborough�̓C�M���X�̒n���j�D���̋ۊ��̖��O�́C20���I�̏����{�茧�̂��鐅�c�ŋN��������̑��Q���C����������w�_�w���ɂ�����蔎�m���������Č������{�ۂ̑�ʔ����ł��邱�Ƃ�˂��~�߂����ƂɗR������D�uDesulfovibrio�v�͋ۑ̂̑��̖��́C�uvulgaris�v�͎햼�C�����āuMiyazaki�v��uHildenborough�v�͋ۊ��̖��̂ŁC�ʏ픭���E�P�����ꂽ�n�����t�����Ă���D�uMiyazaki
F�v�́uF�v�́uformate dehydrogenase�v���ێ����Ă���ۑ̂ł��邱�Ƃ��Ӗ����Ă���C�c�O�Ȃ���I���W�i���̋ۑ̂͌��ݕۑ�����Ă��Ȃ��D���E���ő����̗��_�Ҍ��ۂ��P������C�ۑ̂̃f�[�^�o���N�ɕۊǂ���Ă���D2002�N�ɂ����āCD. v. Hildenborough�݂̂̃Q�m����ǂ�2004�N��D. v. Hildenborough�ɂ��Ă̓Q�m����ǂ��A�����J�Ŋ��������D
�@�@�@�ΐ����C���ł��邱�Ƃ�C�ۑ̂̒P���E���i����������Ƃ��獑���Ɍ����҂����Ȃ��D�������C���Ƃ��Ƃ��̗��_�Ҍ��ۂ̓I�����_�Ɠ��{�̔������w�҂ɂ��Ɨ��Ɍ������i�߂��Ă��Ă���C���{�ł̌����̗��j�͒����D�}�[�J�[�^���p�N���Ƃ��Ă��4�w���E�`�g�N����c3��[NiFe]�q�h���Q�i�[�[�Ȃǂ͂��̗��_�Ҍ��ۂ̂��̂����E���ōł����ڂ���Ă���C�����������镪��Ō�������Ă���D�{�ۑ̂͒n����̃C�I�E�̏z�T�C�N���Ƃ������_�Ŋ��Ȋw�S�ʂ̕��삩��C�܂��n���ɖ��߂������̃p�C�v�̕��H�Ȃǂɂ��W���Ă��邱�Ƃ���y�E���z�H�w�ɂ������b�Ȋw�̕��삩��傢�ɋ�����������Ă���D
�q�h���Q�i�[�[
�@�@�@���ɓd�C��ʂ���Ɛ��f�Ǝ_�f�ɕ������܂��D�t�ɁC���f�Ǝ_�f�����w����������Ɛ������C�����ɔ��d���܂��D���ꂪ�R���d�r�̎d�g�݂ł��D���f�Ǝ_�f��������������ΘA�����d����̂Ŕ��d���u�Ƃ��ė��p�ł��܂��D�܂�C���f�̓G�l���M�[�ɂȂ�킯�ł��D���f��R���Ƃ��ė��p���Ă�������������邾���Ȃ̂ŁC����͋��ɂ̃N���[���G�l���M�[�ł��D20���I�͐Ζ��R���̑�ʗ��p�ɂ����j�傫�Ȗ��ƂȂ��Ă��܂��D���f�͊ԈႢ�Ȃ������̃G�l���M�[�ł���C���f�̗��p�͒n���S�̂̃G�l���M�[�E���������ɖ𗧂��܂��D
�@�@�@�����������f�����������蕪�������肷��\�͂������Ƃ͌Â�����m���Ă��܂����D����ɂ̓q�h���Q�i�[�[�Ƃ����^���p�N���i�y�f�j���d�v�Ȗ������ʂ����Ă��܂��D���̍y�f�����f�̉��w�������ȒP�Ɉ����N�����̂ł��D���̍y�f�̋@�\�i�������J�j�Y���j��m��C��������p����ɂ͗��̍\����m��Ȃ���Ȃ�܂���D����܂Ŏ������́CSPring-8�̕��ˌ�X���𗘗p���ăq�h���Q�i�[�[�̗��̍\������͂��C��������̂��Ƃ��𖾂��Ă��܂����D
[NiFe]�q�h���Q�i�[�[
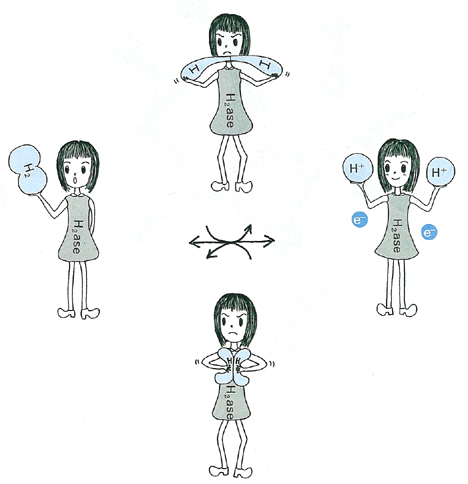
�@�@�@[NiFe]�q�h���Q�i�[�[�͋ۑ̍זE�����O�̃v���g���Z�x���z�߂��Ă���y�f�ŁC�����̍ۂɂ����ăG�l���M�[��ӌn���~���ɐi�߂��ŏd�v�Ȗ�����S���Ă���D���_�Ҍ��ۂ̃q�h���Q�i�[�[�́C�������ʂ̃^�C�v�ɂ��[NiFe]�q�h���Q�i�[�[�C[Fe]�q�h���Q�i�[�[�C[NiFeSe]�q�h���Q�i�[�[��3��ނɕ��ނ���Ă���D
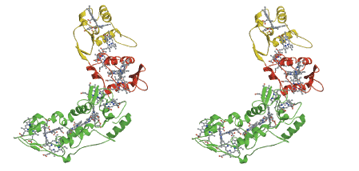
Desulfovibrio vulgaris Miyazaki F���� [NiFe]�q�h���Q�i�[�[�́C[NiFe]�����łȂ�Fe4S4�CFe3S4�CMg�Ȃǂ̋����N���X�^�[�������Ă���D�{�y�f�͕��q�ʖ�29,000�̃��T�u���j�b�g��63,000�̃��T�u���j�b�g�����w�e���_�C�}�[�\�����Ƃ��Ă���D�@�q�h���Q�i�[�[�͎��̂悤�Ȕ�����G�}���Ă���D�����ŋۑ̓��̐����I�d�q�`�B�̂̓`�g�N����c3�ł���.
H2 + �_���^�d�q�`�B�́@�́@2H+ + �Ҍ��^�d�q�`�B�́@+ 2e-
�_���^[NiFe]�q�h���Q�i�[�[�̍\�����
| �@�@�@���݂܂łɁCD. v. Miyazaki F�̎_���^[NiFe]�q�h���Q�i�[�[������\1.8Å�Łi�}-1�j�CDesulfovibrio
gigas�̎_���^[NiFe]�q�h���Q�i�[�[������\2.5Å�ō\����͂���Ă���D�����2��̍y�f��1���\���̑������͍����C3���\�������ɗǂ����Ă����D�������C�������ʂ̍\�������ҊԂňقȂ��Ă����DD. v. Miyazaki F��[NiFe]�q�h���Q�i�[�[�̊������ʂ�Ni���q�ɂ�4�̃V�X�e�C���c��̃C�I�E���q���z�ʂ��C�����̂����C2��Fe���q�ɂ��z�ʂ��ău���b�W���`�����Ă����D�܂�Fe���q�ɂ͔�A�~�m�_�R����4�̔z�ʎq������C���̂���3�{��SO�i�܂���CN�j,
CO, CN���q�Ɠ��肳��Ă���D |
|
�}-1 |
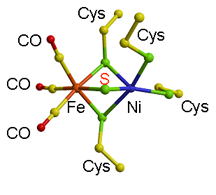
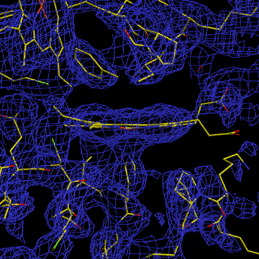
�܂��c��̈�̔z�ʎq�͒P�Ƃ̃C�I�E���q�iS�܂���SH�j��Fe��Ni���u���b�W���Ă��邱�Ƃ��킩�����i�}-2�j������D�Dgigas��[NiFe]�q�h���Q�i�[�[�̔z�ʎq�iCO, CN, CN,
O�܂���OH�j�Ƃ͈قȂ��Ă����D�����̔�A�~�m�_�z�ʎq�̂���2���q���q�z�ʎq��[NiFe]�q�h���Q�i�[�[�̎�Ԃ��邢�͓�������ɂ����Ă��قȂ�i�w�e���j���Ƃ��\�z�����D
 �}-2 �}-2 |
|
 �}-3 �}-3 |
�Ҍ��^[NiFe]�q�h���Q�i�[�[�̍\�����
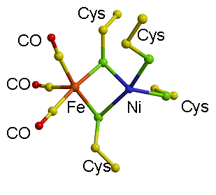
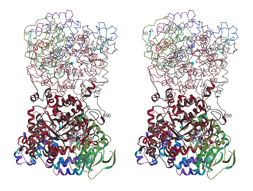
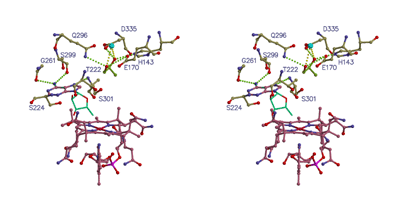
�@�@�@D. v. Miyazaki F �̐��f�Ҍ��^�q�h���Q�i�[�[�̂w�������\����͂��s�������ʁi����\1.4
Å�j�C�_���^�ƊҌ��^�̑S�̍\���ɂ͂قƂ�Ǖω����Ȃ�,�@�قȂ��Ă���_��Ni��Fe���u���b�W���Ă���C�I�E�z�ʎq�̓d�q���x�����ł������D���f�Ҍ�����邱�Ƃɂ���Ă��̔z�ʎq���������ʂ���V���������Ă��邱�Ƃ����炩�ɂȂ����i�}-3�j�D
�@�@�@����܂œ��������ł͓d�q�`�B�̂ƂƂ��ɐ��f�Ҍ�����[NiFe]�q�h���Q�i�[�[���K���������f�����邱�Ƃ��y�f���w�I�Ɍ��o���Ă����D���̗V����������Ă��闰�����f��[NiFe]�q�h���Q�i�[�[1���q������C���悻0.4-0.7���q�ł���ƒ�ʂ��ꂽ�D���̔����ŁC�d�q�`�B�̂�������Η������f�̔����͔��ɒx���C�܂����f�Ҍ�����Ȃ���Η������f�͔�������Ȃ��D�܂�C[NiFe]�q�h���Q�i�[�[�͎_���^���犈���^�Ɉڍs����Ƃ��Ɏ���C�I�E�������o���C����𗰉����f�Ƃ��ėV�����Ă��邱�ƂɂȂ�D�����̎��������Ɩ{���f�Ҍ��^�\����͂̌��ʂ��C�y�f���w�I��@�ɂ�茟�o���ꂽ�������f��Ni-Fe�������S�̃u���b�W�z�ʎq���V���������̂ł��邱�Ƃ��������t������̂ł������D����́C�u���b�W�z�ʎq�̌��q�킪���Ƃ��Ǝ_�f���q�ł͂Ȃ��C�C�I�E���q�ł��邱�Ƃ��Ӗ�������̂ł���D
�@�@�@�ȏ�̌��ʂ���C�q�h���Q�i�[�[�̐��f�������@�\�ɂ����Ă�2�̃T�C�N���C���Ȃ킿�C�������T�C�N���ƐG�}�T�C�N�������݂��C���̃C�I�E�u���b�W�z�ʎq�͊������S�𑼂̔z�ʎq�i�Ⴆ�C�_�f�C���f�Ȃǁj�����邽�߂̖����������Ă���Ƃ�������������D�������T�C�N���ɂ����āC�y�f�����f�ɂ���ĊҌ������ƁC�܂��C���f���������ʂ�Ni�Ɍ������C5�z�ʏ�Ԃł�����Ni�i�V�j��6�z�ʏ��Ni�i�U�j�ɂȂ萅�f�C�I���Ɠd�q����o����D���o���ꂽ�d�q�͓S�C�I�E�N���X�^�[Fe3S4���Ҍ�����̂ɏ�����D�c��̐��f�C�I���̓C�I�E���q�Ɍ������CSH-�ƂȂ�u���b�W���O��āC�������f�Ƃ��ĉ�������D�܂��C��x�C�I�E�u���b�W���Ȃ��Ȃ�ƁCNi��Fe�̊ԂŐ��f�̕��������̐G�}�T�C�N�����m������D���̏�Ԃ́C�ĂуC�I�E��Ni��Fe�̊ԂɁu�W�v������܂ő����D�܂�C�C�I�E�u���b�W�͍זE���̗n�}���̑��̉������␅�_�������犈�����ʂ�ی삷��悤�ɊW�����Ă��āC�G�}�T�C�N�����n�܂�Ƃ��ɂ͈ړ�����悤�ɂȂ��Ă���ƍl���Ă���D
��_���Y�f�����^[NiFe]�q�h���Q�i�[�[�̍\�����
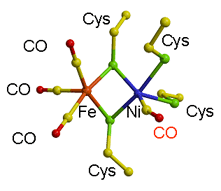
| �@�@�@����܂Ńq�h���Q�i�[�[�̐��f�������̏�����Ԃɂ����āC���f�Ƃ̔������ʂ�Fe�ł���̂�Ni�ł���̂����炩�ł͂Ȃ������D���f�̏����������ʂ����肷�邽�߁C���f�̋����j�Q�܂ł����_���Y�f�������������u��_���Y�f�����^�q�h���Q�i�[�[�v�̋����}�����������X�������\����͂��s�����D�܂��C��_���Y�f�����Ǝ˂ɂ��𗣂��������𗣌^�q�h���Q�i�[�[��X�������\����͂��s�����D�O�����������ꂽ��_���Y�f�́CFe�ł͂Ȃ�Ni�ɔz�ʂ��C���̌����l���͒����I�ł͂Ȃ��C�Ȃ����Ă���i135�`160���j���Ƃ����炩�ƂȂ����i�}-4�@�{CO�����^�q�h���Q�i�[�[�̉�͌��ʂł�Fe�ɔz�ʂ��Ă���2���q���q�͑S��CO�ł���Ɖ��肵�Ă���j�D |
|
�}-4 |
�@�@�@�܂��C��_���Y�f��100K�ł͐��f�ɂ��ȒP�ɂ͒u������Ȃ����C�������F���ē����ɐ��f�Ҍ����邱�Ƃɂ���ėe�Ղɉ𗣂��邱�Ƃ����炩�ƂȂ����D�{�\����͂̌��ʁC�j�Q�܁i��_���Y�f�j��Cys546�̃C�I�E���q�̊Ԃɂ킸���ȓd�q���x���c���Ă���C����͈�_���Y�f�ɂ��j�Q�v���Z�X�̓r���Ő��f���g���b�v����Ă�����̂ƍl�@�ł����D�܂��C��_���Y�f�����^�ƌ��𗣌^��Fo�|Fo�}�b�v���v�Z�������ʁC
Ni���q��Cys546�̃C�I�E���q�̓d�q���x�݂̂��ړ����邱�Ƃ����炩�ƂȂ����D���̃C�I�E���q��[NiFeSe]�q�h���Q�i�[�[�ł́CSe�ɒu��������Ă��錴�q�ł������D�����̌��ʂ��CNi���q��Cys546�̃C�I�E���q���C���f�̏������������ɏd�v�Ȗ�����S���Ă���ƌ��_�����D
�@�@�@���݂͎_���^�q�h���Q�i�[�[�Ɍ��o����Ă����̌^�iNi-A�^��Ni-B�^�j�̈Ⴂ���\�����w�I�Ɋ��S�ɖ��炩�ɂ���ׂ������𑱂��Ă���D
�@�@�@�{�����͐É���w�̔��ؒB�F�C���s��ȑ�w�̜A�c�r�C�h�C�c�E�}�b�N�X�v�����N��������Lubitz���Ƃ̋��������v���W�F�N�g�ł���D
HypF�^���p�N��
�@�@�@�^���p�N���͓Ǝ��̗��̍\�����`��邱�Ƃɂ�菉�߂Ă��̋@�\������D�q�h���Q�i�[�[�Ƃ����^���p�N���͔������ɑ��݂���y�f�Ő����I�@�\�͐��f��ӂɊ֗^�ł���D�ߔN�C�q�h���Q�i�[�[�̐��n���ɊW�����`�q�Q���V���ɔ������ꂽ�D������hypA�C hypB�C hypC�C hypD�C hypE�C hypF�ƌĂ�Ă���DhypF ���|���HypF�͕��q�ʖ�82,000�̑��@�\�^���p�N���ŁC3�̃h���C���������Ă���D�����́C���ꂼ��A�V���z�X�t�@�^�[�[�h���C���CO-�J���o���C���h���C���C������DnaJ�V���y�����Ɠ��l��2��zinc
finger���`�[�t�����h���C���ł���D
�@�@�@�q�h���Q�i�[�[�̊������ʂ͔��ɕ��G�Ȍ`�����Ă���CNi���q��Fe���q�ō\������Ă���D�܂��C��ʓI�ɂ͐����ɑ��ēŐ�������CO���q��CN���q��Fe���q�Ɍ������Ă���D�����̏����q���q�h���Q�i�[�[�Ɏ�荞�܂�邽�߂̔����́CHypF�ɂ��J���o���C���z�X�t�F�C�g��CO��CN�ɓ]������C�ŏI�I�Ƀq�h���Q�i�[�[�̊������ʂɂ���Fe���q�ɔz�ʂ����Ƃ����ߒ��ނƍl�����Ă���D���̂悤�Ƀq�h���Q�i�[�[�̐��n���͕��G�ȋ@�\�������Ă���Ɨ\�z����Ă��邪�C
���݂܂Ō��q���x���ł̉�͂Ɋ�Â������@�\�͉𖾂���Ă��Ȃ��D�������́C����HypF�̗��̍\���𖾂炩�ɂ��邱�Ƃɂ��C�q�h���Q�i�[�[�̐��n���̕��q�@�\���𖾂��邱�Ƃ�ڎw���Ă���D
�@�@�{�����͓Ɨ��s���@�l�E�_�Ɛ��������������̕x�R����Ƃ̋��������v���W�F�N�g�ł���D

DsrD�^���p�N��
| �@�@�@DsrD�͈����_�Ҍ��y�f�iDsrA2DsrB2�j���R�[�h�����`�q�Q�idsr�I�y�����j����dsrD���甭�������^���p�N���ł���i�}-1�j�D�����_�Ҍ������Ɛ[���ւ�肪����ƍl�����Ă��邪�C���̋@�\�͂܂��悭�������Ă��Ȃ��D�܂��C�A�~�m�_�z��͗ގ��^���p�N���͌������Ă��Ȃ������D��X��Desulfovibrio vulgaris Hildenborough�R���EDsrD�̗��̍\�����炻�̐����I�@�\��ސ����ׂ��C��������\X�������\����͂�ڎw�����D |
|
�}-1 |
 |
 |
| �}-2 |
�}-3 |
|
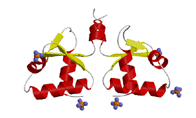
�@�@�@�{DsrD��78�A�~�m�_�c���Ȃ镪�q��8700�̔�r�I�����ȃ^���p�N���ł���D2000�N�ɗ��_�A�����j�E���𒾓a�܂Ƃ��Č������ɐ������C2003�N��1.2Å����\�łw�������\�����ɐ��������D���̕��q�̐����I�@�\�́u�����_�C�I���̃g�����X�|�[�^�v�ł͂Ȃ����Ɠ����l�����Ă����D�������C����̍\����͂���DsrD��winged-helix���`�[�t�������Ƃ����炩�ɂȂ����i�}-2
DsrD�̌�������2�ʑ̍\���C2�ʑ̂�����5�̗��_�C�I�����������Ă���j�D�]����DsrD��DNA�����^���p�N���ł���\�������������DDsrD��winged-helix���`�[�t�̗��̍\���l�̃��`�[�t��������DNA�����^���p�N���Əڍׂɔ�r�������s�����Ƃ���CdsRNA
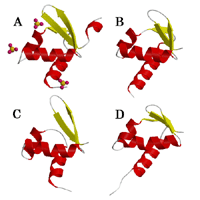
adenosine deaminase (ADAR1)��Zα�h���C���ƍł��ގ����Ă��邱�Ƃ��킩�����i�}-3
(A)DsrD�C(B)ADAR1��Zα�h���C���C(C)SmtB�C(D)Histone
H5�j�DZα�h���C����Z-DNA�����^���p�N���ł���ƕ���Ă���D�����̍\�����w�I�Ȓm��������DsrD�Ƒ��ݍ�p������B-DNA��Z-DNA�̒T�����s���Ă��邪�C�����_�ł͌������Ă��Ȃ��D
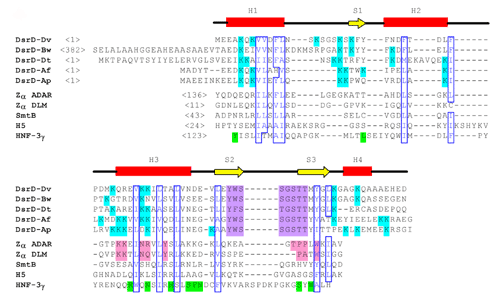
�@�@�@�܂��C�{�\�����w�I�Ȍ����̌��ʁCDsrD���܂߂�winged-helix���`�[�t������DNA�����^���p�N���̓A�~�m�_�z��ɂ͂����肵���ގ��_�͖������C���̃��`�[�t�\�����ێ����邽�߂ɑa�����c��i���Ƀ��C�V���j���d�v�Ȗ�����S���Ă��邱�Ƃ����炩�ƂȂ����i�}-4�@
DsrD�Ɨގ��\���������q�̃A�~�m�_�z��̔�r�j�D
�}-4
�@�@�@�ŋ߁C�����_�Ҍ��y�f�̂ЂƂ̃T�u���j�b�g�iDsrB�j��DsrD��Z�����Ĕ�������ۊ������������D�����DsrD�������_�Ҍ��������邢�͍y�f�̓]�ʁE�|��Ɋ֗^���邱�Ƃ��������Ă���D����CRNA���܂߂�DsrD�Ƒ��ݍ�p����j�_�̒T���𑱂��C���̋@�\�𖾂炩�ɂ��邽�߂̎������s���K�v������i�}-5�@���낢���DNA�����^���p�N��
��B-�����Z-DNA�j�D
�@�@�@�{�����̓J���K���[��w��Voordouw���y�ы�B�H�Ƒ�w�̎M�䌤�����Ƃ̋��������v���W�F�N�g�ł���D |
|
�}-5 |

�����q�ʃ`�g�N����c
�@�@�@�����q�ʃ`�g�N����c�iHMC = high-molecular weight cytochrome
c�j�͂��悻550�̃A�~�m�_�c���Ȃ�w���^���p�N���ł���D�É���w�̔��ؒB�F�����_�Ҍ��ۂɓ��ٓI�ȍ����q�ʂ̂�-�^�w���E�`�g�N�����Ƃ��Ĕ������C���̌�C���������̔���������q�ʁi��65,000�j�C�z���X�y�N�g���C���d�_�ipI=9.1�j�C�w���̌��i16�j�Ȃǂ����(Higuchi
et al., 1987)�D�Ҍ��^���q�̓��ٓI�ȋz���̃s�[�N�ʒu�́Ca=532,
b= 532, g=410 nm�ł���D1�{�̃|���y�v�`�h����16�̃w����L����Ƃ����͓������^�I�Ɍ����Ă����D������Voordouw���ɂ��Desulfovibrio vulgaris Hildenborough����HMC�̃I�y��������ǂ���C16�́u-CXXCH-�v�Ƃ����ꎟ�\�����`�[�t�������Ƃ��m�F���ꂽ�D���q���ɂ����邱�̃��`�[�t�̐��Ɣz��l������HMC�̗��̍\����4�̃`�g�N����c3�̍\���h���C������\������Ă���ƍl����ꂽ�D
�@�@�@��X�̌������ł�D. v. Hildenborough�����Miyazaki����HMC�ɂ��č\�����w�I�����𑱂��Ă��Ă���D�@���^���p�N���̗��̍\���͊��ɉ�͂���Ă�
��C�܂��Ȃ��_�����\�����\��ł���DHildenborough��HMC�̕��q�S�̂̍\����}-1�Ɏ����D�\�z���ꂽ�Ƃ���C�{���q�̍\���̓`�g�N����c3��4-�w���\���h���C������{�Ƃ��č\������Ă����D�h���C��-I�i�w��-1�C2�C3�j�͉��F�C�h���C��-II�i�w��-4�C5�C6,
7�j�͐ԐF�C�����ăh���C��-III�i�w��-8, 9,
10, 12 ����� 11�j��-IV�i�w��-13�C14�C15,
16�j�͗ΐF�Ŏ����Ă���D �h���C��-III��-IV��9Hcc (nine-heme cytochrome c)��2�̍\���h���C���Ɣ��ɗǂ������\���������Ƃ����炩�ƂȂ����D�w���ƃh���C���̔ԍ���N�����珇���������Ă���D
|
|
|
| �}-1 |
|
�}-2 |
�@�@�@�h���C��-IV�Ɋ܂܂��w��-15��5�z�ʌ^�ł���CEPR�X�y�N�g����蓯�肳�ꂽ���X�s����Ԃ̃w���͂��̃w��-15�ł���ƍl������D�܂��C��ʂ̑�6�z�ʎq�t�߂ɂ̓C�\���C�V�������݂��Ă���(�}-2)�D
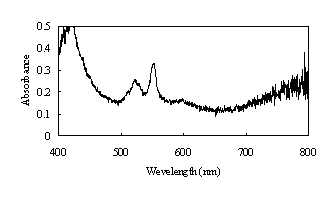
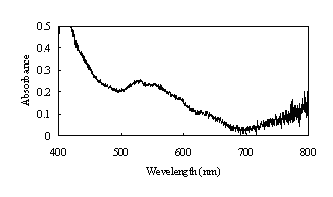
�@�@�@�ŋ߂̉�X�̌�������C����HMC�̌�����X��������ɂ�����X���̏Ǝ˂ɂ��_���^�i�}-3a�j����Ҍ��^�i�}-3b�j�Ɉڍs���Ă������Ƃ����炩�ɂȂ����D�]���ăV���N���g�������ˌ��ɂ�鍂�P�x��X����p���ĉ�͂��ꂽ���̎�̃`�g�N�����̗��̍\���͎_���^����Ҍ��^�ւ̑J�ڏ�Ԃ����Ă���\�������������D
 |
 |
| �}3-a |
�}3-b
|
�@�@�@HMC�I�y�����ɂ͂������̃^���p�N�����R�[�h����Ă��邱�Ƃ��킩���Ă���D���_�Ҍ��ۂ͖��̓��O�œd�q�̂��Ƃ���s�����߁C���̖�����S���V�X�e�������ɑ��݂���͂��ł���DHMC�͂��̂悤�Ȗ����O�̓d�q�`�B���s���Ă���̂ł͂Ȃ����ƍl������D�܂��C16���̃w��������HMC���������ꂽ�ێ�͗��_�Ҍ��ۂ̒��ł������Ă���D����͌��C���ۂ̐i���̖ʂ������ϋ����[�����Ƃł���D
�@�@�@�{�����͑���w�̈��v�Ì������Ƃ̋��������v���W�F�N�g�ł���D

�e�l�m�����^���p�N��
�@�@�@�e�l�m�����^���p�N��(FMN-binding protein)�́C���_�Ҍ���Desulfovibrio vulgaris Miyazaki F�̃t�F���h�L�V����`�q�̃N���[�j���O���s���Ă��鎞�ɖk�����ɂ����R�������ꂽ�^���p�N���ł���D���q1������1���q�̂e�l�m��⌇���q���Ƃ��ĕێ����Ă���D122�A�~�m�_�c��琬�镪�q�ʂ�13,700�̃^���p�N���ŁC���ݕ���Ă���FMN�����^���p�N���̒��ł͍ł����������̂ł���D�����@�\�͌��ݕs���ł��邪�C���̂e�l�m�^���p�N�����_���Ҍ��ɗ��p����Ă��邱�Ƃ��炱�̃^���p�N�������l�̐����������Ă�����̂ƍl�����Ă���D���������ł͂e�l�m�����^���p�N���̂w�������\����͂�ʂ��āC���̋@�\�ƍ\���ɂ��Ă̒m���邱�Ƃ�ړI�Ƃ��Č������Ă���D
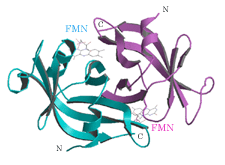 |
|
 |
| �}-1 |
|
�}-2
|
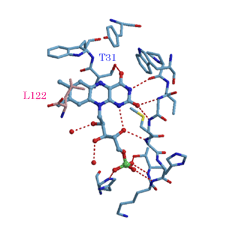
�@�@�@�e�l�m�����^���p�N���͗n�t���ł͒P�ʑ̂ł��邱�Ƃ�����Ă��邪�C�w�������\����͂̌��ʂ���C�������ɂ����Ă͓�ʑ̂��`�����Ă��邱�Ƃ����炩�ɂȂ����i�}-1�j�D�e�l�m�͂����2�ʑ̂̕��q�Ԃɋ��܂�Ă���C2�ʑ̒��̈���̕��q(���F)�Ɠ��ٓI�ɐ��f�������C�܂������̕��q�i�ԐF)�Ƃ��a�������ݍ�p�����Ă����D�����i�ԐF�j�̕��q�̂b���[�ɂ���Leu122�́C�e�l�m�̃�-xylene���ʁi�a���������j���n�}�̈�ɘI�o����̂�h���悤�ȍ\�����Ƃ��Ă����D������CLeu122�͂e�l�m�̉𗣒萔�̌����╪�q�̎_���Ҍ��d�ʂɊW���Ă���̂ł͂Ȃ����ƍl�@�����D
�@�@�@���݁CFMN�^���p�N����Leu122��ψق������^���p�N���́iL122Y�CL122K�CL122E�jX�������\����͂��s���CL122�̖����Ȃǂɂ��ďڍׂɌ������ł���D
�@�@�@�{�����͑��s����w�̖k�����瓙�Ƃ̋��������v���W�F�N�g�ł���D

���u���h�L�V��
| �@�@�@���u���h�L�V���́C�����̍ۂɌ��o����Ă���^���p�N���̂ЂƂł���D�`�g�N����c-553�Ɠ��l�ɏ����ȃ^���p�N���ŁC�A�~�m�_�c��͂��悻50-60�C���q��6000���x�̑傫���ł���D����̓w���^���p�N���ł͂Ȃ����q���Ɍ��q���Fe��d�q�`�B�̊������S�Ƃ��Ċ܂ށD���_�Ҍ��ۂ̃��u���h�L�V���́C���u���h�L�V��-�_�f�E�_���Ҍ��y�f�iROO�j�̐����I�d�q�`�B�̂ł���ƍl�����Ă���D |
|

�}-1 |
�@�@�@�{�������ł͗��_�Ҍ��ہEDesulfovibrio vulgaris Miyazaki F�R���̃��u���h�L�V����X�������\����͂��s�����D���̕��q��1���q����1��Fe���q��2�̃V�X�e�C���c����̃C�I�E���q�ŕێ����Ă���D
���������ł�1998�N�ɖ{�^���p�N����3�������̍\�����𖾂����i�}-1�j�D�����͈قȂ��������2�̏��n�ɂ��ē���ꂽ�D�{���̍\���ƃA�~�m�_�z��̗ǂ�����Hildenborough���̂��̂Ƃ��r���邱�Ƃɂ��C���u���h�L���̕\�ʍ\�����ǂ̂悤�Ɍ������Ɋ֗^���Ă��邩�Ȃǂɂ��ďڂ����c�_���邱�Ƃ��ł����D
�@�@�@�{�����͐É���w�i�����j�̔��،������Ƃ̋��������v���W�F�N�g�ł���D

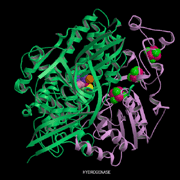
�`�g�N����c3
| �@�@ �`�g�N����c3�́C�����_�Ҍ��y�f�iDesulfoviridin�j�ƕ��ї��_�Ҍ��ۂ̃}�[�J�[�^���p�N���Ƃ݂Ȃ���Ă������q�ł���D�ے��ł͐��f�_���Ҍ��y�f�E[NiFe]�q�h���Q�i�[�[�̓��ٓI�d�q�`�B�̂Ƃ��������I�Ȗ�����S���Ă���D�`�g�N����c3�͕��q��14000���x�̉���^���p�N���ŁC�P���q����4��c�^�w�����܂ށD4�̃w���̕��ς̎_���Ҍ��d�ʂ͔�r�I�Ⴂ�l�i��-300mV�j�ł���C�܂��ő����ɂ��ăq�h���Q�i�[�[������������ƍ����f�����ɂ����Ĕ��ɍ����d�������������Ƃ���l�X�ȕ������w�I���������ׂ��Ă��Ă���D |
|
�}-1 |
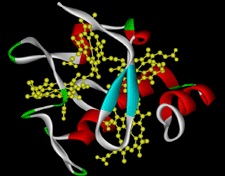
�@�@�@��X�͂���܂ł�Desulfovibrio vulgaris Miyazaki�i�}-1�j�����Hildenborough�R���̎_���^�`�g�N����c3�̗��̍\������͂��Ă����D����܂łɐ���ނ̃`�g�N����c3 �̌����\��������Ă���C�����̈ꕔ�̓A�~�m�_�z��̑����������ɒႢ�ɂ��ւ�炸4�̃w���̑��Δz�u�₻�����芪���卽�̐܂ꂽ���ݍ\���͗ǂ����Ă��邱�Ƃ��m���Ă���D
�@�@�@���̍\���Ɋ�Â��l�X�ȓd�C���w�I����ѕ����w�I�Ȍ������s���C�`�g�N����c3�ɂ�������قȕ������w�I�����͂��̃w���̑��Δz�u�Ƃ����̋߂��ɑ��݂������ꂽ���̃A�~�m�_�����␅���q�̍\���ɗR������ƍl�����Ă���D�ŋ߁C����w�̏��V�E���v�Ó���Shewanella oneidensis ���h��ɗp�����n�Ń`�g�N����c3�̑�ʔ����n�̍\�z�ɐ��������D����𗘗p���ĕ��q�̓d�q�`�B�ɏd�v�Ɨ\�z�����c��ɕψق�^���Ă����̎_���Ҍ��d�ʂ̕ω��𗧑̍\�����痝�����ׂ������\����͂��s���Ă���D
�@�@�@�{�����͑���w�̈��v�Ì������̋��������v���W�F�N�g�ł���D

�`�g�N����c-553
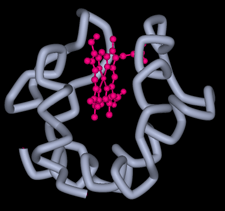
| �@�@�@�`�g�N����c-553�́C�����̍ۂɌ��o����Ă���w���^���p�N���̂ЂƂł���D�A�~�m�_�c��͂��悻70-80�C���q��8000���x�̔�r�I�����ȃ^���p�N���ŕ��q����1�̂�-�^�w����ێ����Ă���D�w���S�̃^���p�N���z�ʎq�̓q�X�`�W����Nε2����у��`�I�j����Sδ���q�ł���D�{�������ō\����͂����`�g�N����c-553�͗��_�Ҍ��ہEDesulfovibrio vulgaris Miyazaki F�R���̂��̂ō����q�ʃ`�g�N����c��`�g�N����c3�Ƃ̊Ԃœd�q�`�B��S���Ɨ\�z����Ă��邪�����I�Ȗ����ɂ��Ă͖����s���ȓ_�������D |
|
�}-1 |
�@�@�@���������ł�1980�N�ɖ{�^���p�N���̌������ɐ��������D�{�����͓��d�_�����ɍ����ipI=10.5�j�^���p�N���ł��邱�Ƃ���C�^���p�N���n�t��pH���A�����j�A�̏��C�g�U�ɂ�蓙�d�_�߂��܂ŏ㏸�����Č��������s�����D����ꂽ��������d���q���`�u���̂邱�Ƃ�����ł��������C1991�N�ɍ��G�l���M�[�����w�������E���ˌ��{�݁i���E���G�l���M�[�����팤���@�\�E�����Ȋw�������j�̕��ˌ�X����p���āCFe���q�̑��g���ُ핪�U�@�ɂ�肻��3�������̍\�����𖾂����i�}-1�j�D�{�\����͂̌��ʁC���q�ʂ̏�����1�w���`�g�N����c�̕��q�i���ɂ��Ă̒m���\�����D
�@�@�@�{�����͐É���w�i�����j�̔��،���������ё���w�i���݁j�̒��쌤�����Ƃ̋��������v���W�F�N�g�ł���D
 

�W�I�[���f�q�h���^�[�[
| �@�@�@�r�^�~��B12�͐��̓��Ɏ�荞�܂��ƕ�y�f�^�ł���A�f�m�V���R�o���~���܂��̓��`���R�o���~���ɕϊ������D�O�҂ِ͈����C�E���C�]�ځC�Ҍ��Ȃǂ̔����Ɋ֗^���C��҂�C1��ӂɊ֗^�����`�I�j���V���^�[�[�̕�y�f�Ƃ��ċ@�\����D�R�o���~���ɂ̓R�o���g�C�I�������݂��C���̏���z�ʎq�ɂ̓A�f�m�V����i�A�f�m�V���R�o���~���j�܂��̓��`����i���`���R�o���~���j���z�ʂ��邱�Ƃɂ���ăR�o���g�|�Y�f�����iCo-C�����j���`������DCo-C�����̉t�I���ɂ���ď���z�ʎq��������ƂȂ��Ɣ������邪�R�o���g�C�I���͔����ɒ��ړI�ɂ͊֗^���Ȃ��D |
|
�}-1 |
�@�@�@��X�̃O���[�v�̓r�^�~��B12����y�f�Ƃ��č�p���郁�J�j�Y���𖾂炩�ɂ��邽�߂ɃA�f�m�V���R�o���~�����y�f�Ƃ���W�I�[���f�q�h���^�[�[��X�������\����͂��s���i�}-1�j�D������^�Ɣ��^�̗��̍\���̍\���ω��ƃA�f�m�V���R�o���~���̃A�i���O�ł���A�f�j�j���y���`���R�o���~���Ƃ̕����̗̂��̍\������Co-C�����̉��̃��J�j�Y�������炩�ɂȂ����D
�@�@�@�܂�����݉��ł̃A�f�j�j���y���`���R�o���~���Ƃ̕����̂̍\����͂̌��ʁC����z�ʎq�ɂ����ăA�f�m�V���R�o���~���Ƌ��ʂ̉��w�\�������A�f�j���������ʂ̍\�������m�ƂȂ����i�}-2�j�D�A�f�m�V����̗��̍\�����A�f�j�����ʂɂ����ďd�ˍ��킹���Co-C�Ԃ̍ŒZ������3.11���ƂȂ�C�A�f�m�V���R�o���~���P�Ƃł̌����\���i�}-3�j�ɔ�ז�1.1��
�����Ȃ��Ă����i�P�Ƃł�1.98���j�D���̍��̓A�f�m�V���R�o���~���ɂ�����Co-C�Ԃ̋�������1.1���Ȃ邱�Ƃ��Ӗ����Ă���D���̌��ʁC�������Ԃł�Co-C��������Ă�����̂ƍl������D�܂��C����^�̍\���ł�Co-C�����̋�������2.98���ł������D�܂����^�̕����A�f�m�V���R�o���~���P�Ƃł̌��������ɂ��߂����Ƃ����������D
�}-2
�}-3 |
�@�@�@�ȏ�̌��ʂ���A�f�m�V���R�o���~�����y�f�Ɍ��������Co-C��������1.0�������L����C���₷���Ȃ�ƍl���邱�Ƃ��ł���D�z���y�f�Ɋ����������Ƃ���ɖ�0.1�������L����邱�Ƃ�Co-C�����������̂ƍl������D�܂��y�f�S�̂̍\���ɒ��ڂ���ƁC������^�Ɣ��^�̗��̍\���̍\���ω����������ɂ��Co-C�����̉��ɂ͍y�f�̍\���ω����������Ƃ����炩�ƂȂ����D
�@�@�@�{�����͉��R��w�̌ՒJ�������Ƃ̋��������v���W�F�N�g�ł���D
�֘A���镶��
Shibata, N., Masuda, J., Morimoto, Y., Yasuoka,
N., and Toraya, T. (2002) Substrate-induced
Conformational Change of a Coenzyme B12-Dependent
Enzyme: Crystal Structure of the Substrate-Free
Form of Diol Dehydratase, Biochemistry, 41, 12607-12617.
Masuda, J., Shibata, N., Morimoto, Y., Toraya,
T. and Yasuoka, N. (2001). Radical production
simulated by photo-irradiation of the diol
dehydratase-adeninylpentylcobalamin complex.
J. Synchrotron Rad, 8, 1182-1185.
Masuda, J., Shibata, N., Morimoto, Y., Toraya,
T. and Yasuoka, N. (2000). How a Protein
Generates a Catalytic Radical from Coenzyme
B12: X-ray Structure of Diol Dehydratase-Adeninylpentylcobalamin
complex. Structure, 8, 775-788.
Shibata, N., Masuda, J., Tobimatsu, T., Toraya,
T., Suto, K., Morimoto, Y. and Yasuoka, N.
(1999). The New Mode of B12 Binding and Direct
Participation of Potassium Ion in Enzyme
Catalysis: X-ray Structure of Diol Dehydratase.
Structure, 7, 997-1008.
Masuda, J., Yamaguchi, T., Tobimatsu, T.,
Toraya, T., Suto, K., Shibata, N., Morimoto,
Y., Higuchi, Y. and Yasuoka, N. (1999). Crystallization
and Preliminary X-ray Study of Two Crystal
Forms of Klebsiella oxytoca Diol Dehydratase-Cyanocobalamin Complex.
Acta Cryst., D55, 907-909.
��������, �ēc���� (2001). ���W�J�������̏����ߒ��̍\���������w�FB12��y�f�֗^�W�I�[���f�q�h���^�[�[�̕��ˌ��ɂ�錤��,
��������, 236, 201-204
�ēc����, �������� (2000). �V�K�̃r�^�~��B12�����l�������W�I�[���f�q�h���^�[�[�̗��̍\���ƕ�y�f�������@�\,
���{�����w�, 42, 354-360

�G�^�m�[���A�~���A�����j�A���A�[�[
�H����

�������ۗR���`�g�N���� c'
�@�@�@�`�g�N����
c'�iCytochrome c'�j�͌������ہC�E���f�ۂȂǂɑ��݂��C�w�� c ���T�u���j�b�g������P�܂ޕ��q�ʖ� 14,000
�̒`�����ł���D�ʏ�`�g�N���� c' �̓z���_�C�}�[�Ƃ��đ��݂��邪�CRhodobacter palustris �R���̃`�g�N���� c' (RPCP) �̓��m�}�[�Ƃ��đ��݂���D�܂��CChromatium vinosum (CVCP) �ł̓w���ɔz�ʎq���������邱�Ƃɂ�胂�m�}�[�ɉ𗣂��邱�Ƃ��m���Ă���D
�@�@ |
|
�}-1 |
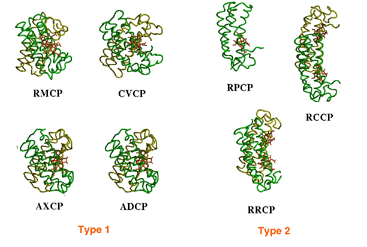
�@�@�@��X�̌����O���[�v�ł́C����܂łɗn�t���Ń��m�}�[�ƃ_�C�}�[�����݂���
Rhodobacter capsulatus �̃`�g�N���� c' (RCCP) �̔z�ʎq���݉��y�є݉��ł̂w�������\����͂ɐ������C�z�ʎq�����ɂ��_�C�}�[�𗣂̃��J�j�Y���ɂ��ĕ����i�}-1�j�D�{�����̖ړI�̓��m�}�[�ő��݂���
RPCP �̗��̍\�����w���\����͂Ŗ��炩�ɂ��C�`�g�N����
c' �̎l���\�������肷��v�����𖾂��邱�Ƃł���D
 |
| �@�@ |
�}-2 |
�@�@�@�`�g�N���� c' �� Four-helical bundle ���`�[�t�iHelix
A-D�j�������C�e�T�u���j�b�g��Helix A�� Helix
B�Ō`�������ʁiA-B�ʁj�ŐڐG���邱�Ƃ��m���Ă���D����܂łɉ�͂��ꂽ�`�g�N���� c' �ł́C�ڐG�ʂɑa�����c��������݂��C�����̎c��ɂ��a�����ݍ�p���_�C�}�[�`���ɏd�v�ł��邱�Ƃ�����Ă���i�}-2����ѐ}-3�j�D����CRPCP
�ł̓��W����4�C�����ăA�X�p���M���_2�C�O���^�~���_��1�ƁC�d�ׂ����������������݂��邱�Ƃ����炩�ƂȂ����D�\�ʓd�ׂ̕��z�ׂ��Ƃ���C���ɔ��A-B�ʂɓd�ׂ������W�܂��Ă����DRPCP
�ɂ����Ă� A-B �ʂɓd�ׂ��������݂��邱�Ƃ���C���̖ʂ̓d�ׂ̗ʂƕ��z���`�g�N����
c' �̃_�C�}�[�ƃ��m�}�[�̑��ݔ�ɑ傫���e�����Ă�����̂ƍl������D
 |
| �@�@ |
�}-3 |
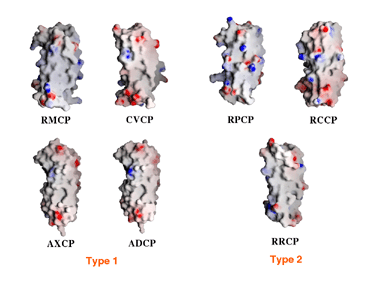
�@�@�@�`�g�N���� c' �̃_�C�}�[�\���͐ڐG�ʂɑ��݂���c��̑g���ɂ���ē����Â�����i�}-2����ѐ}-3�j�DType
1�͑a�����c��������݂��C����ɋ߂��Ȃ�悤��X�^�̃_�C�}�[�\�����Ƃ�DType
2�͓d�ׂ����c��������݂��CType 1�ɔ�׃��m�}�[�\�����Ƃ�₷���D�܂��C2�̃T�u���j�b�g�̓A���`�p�������\���ɋ߂��Ȃ�X���ɂ��邱�Ƃ��킩�����D
�@�@�@�{�����͕č��E�A���]�i��w��Cusnovich�������Ƃ̋��������v���W�F�N�g�ł���D

������������q�^���p�N��
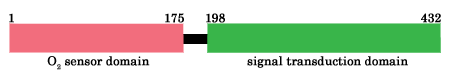
�}-1
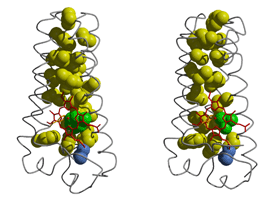
�@�@�@������������q�͌͑��ۂ̎_�f�ɑ��鑖��������n�ɂ����Ď_�f�Z���T�[�Ƃ��ċ@�\����^���p�N���ł���D�_�f�̃Z���T�[�h���C���Ƃ��̃V�O�i����ϊ��E�`�B����g�����X�f���[�T�[�h���C���̓�̗̈�������Ă���i�}-1�j�D
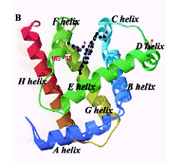
| �@�@�@�_�f�̃Z���T�[�h���C���͕⌇���q���Ƃ��ăw����L���C���̕����̈ꎟ�\���̓~�I�O���r���i�}-2�j�ƍ�����������������N���̈�ɂ͗]���̒����z������D���̂悤�ȋC�̕��q�Z���T�[�^���p�N���Ɋւ��錤���͐��E�I�Ɍ��Ă��n�܂�������ł���C�����̕��q�@�\�ɂ��Ă͂قƂ�ǂ킩���Ă��Ȃ��D��X�́C���̍\����肻�̕��q�@�\�̉𖾂��s�����Ƃ�ړI�ɁC���݂͌����������݂Ă���D |
|
�}-2 |
�@�@�@�{�����͉��荑�����������@�\�E�����o�C�I�T�C�G���X�Z���^�[�̐쌤�����Ƃ̋��������v���W�F�N�g�ł���D
|
|